データ分析をしていると、「数万行のデータなのに集計が遅い」「メモリ不足でエラーが出る」といった問題に直面することがあります。
実は、これらの問題の多くは、データ型を適切に設定するだけで解決できることをご存知でしょうか。
今回は、Pandasのgroupbyという集計機能を簡単に高速化する方法を、ざっくり説明します。
難しい理論は後回しにして、まずは実際に手を動かしながら、どのくらい速くなるのかを体感してみましょう。
Contents
- Pandas groupby の基礎を理解しよう
- そもそもgroupbyとは何か
- 複数の条件でグループ化する
- なぜgroupbyが遅くなるのか
- 原因1:文字列のまま処理している
- 原因2:存在しないグループも処理している
- 原因3:結果を並べ替えている
- 原因4:同じgroupbyを何度も実行している
- 実際に高速化を体験してみよう
- ステップ1:検証用データの準備
- ステップ2:最適化前の処理速度を測定
- ステップ3:データ型を最適化して再測定
- カテゴリ型が効果的かどうかを判定する方法
- 大規模データを扱うときのテクニック
- さらに高速化するためのテクニック
- テクニック1:ベクトル化による高速化
- テクニック2:複数集計は1回で(Named Aggregation)
- テクニック3:PyArrowバックエンドの活用(Pandas 2.0以降)
- まとめ
Pandas groupby の基礎を理解しよう
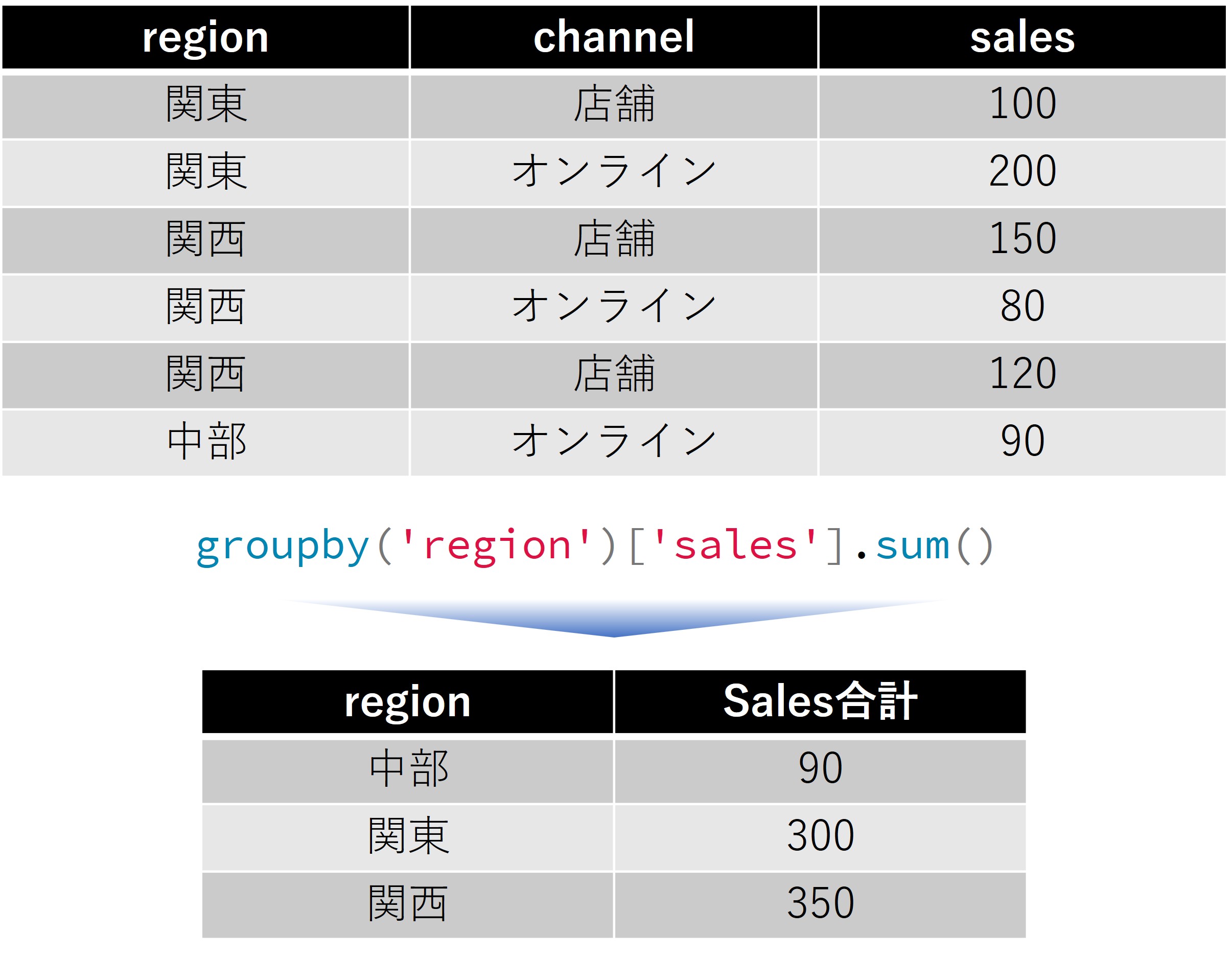
そもそもgroupbyとは何か
groupbyは「グループバイ」と読みます。これは、データを特定の条件でグループに分けて、それぞれのグループごとに計算を行う機能です。
たとえば、全国の売上データがあったとき、「地域ごとの売上合計を知りたい」という場合を考えてみましょう。
関東の売上、関西の売上、中部の売上…と地域別に分けて計算する必要がありますね。
このような「グループに分けて集計する」作業を簡単に実現してくれるのがgroupbyです。
では、小さなサンプルデータを作り、どのような挙動をするのか見てみます。
まずは、サンプルデータを作ります。
以下、コードです。
import pandas as pd
# まずは6行だけの小さなデータで試してみます
df_sample = pd.DataFrame({
'region': ['関東', '関東', '関西', '関西', '関西', '中部'],
'channel': ['店舗', 'オンライン', '店舗', 'オンライン', '店舗', 'オンライン'],
'sales': [100, 200, 150, 80, 120, 90]
})
# データの中身を確認
print(df_sample)
以下、実行結果です。
region channel sales 0 関東 店舗 100 1 関東 オンライン 200 2 関西 店舗 150 3 関西 オンライン 80 4 関西 店舗 120 5 中部 オンライン 90
このデータには、地域(region)、販売チャネル(channel)、売上(sales)の3つの列があります。
ここで「地域ごとの売上合計」を計算してみましょう。
以下、コードです。
# 地域ごとの売上合計を計算
result = df_sample.groupby('region')['sales'].sum()
print(result)
以下、実行結果です。
region 中部 90 関東 300 関西 350 Name: sales, dtype: int64
groupby('region')で地域ごとにグループを作り、['sales']で売上列を選択し、.sum()で合計を計算しています。
関東の2つのデータ(100と200)が合計されて300になっているのがわかりますね。
複数の条件でグループ化する
実際の業務では、「地域別かつチャネル別」のように、複数の条件でグループ化することがよくあります。
以下、コードです。
# 地域×チャネル別の平均売上を計算 # as_index=Falseを付けると、結果が見やすい表形式になります result = df_sample.groupby(['region', 'channel'], as_index=False)['sales'].mean() print(result)
以下、実行結果です。
region channel sales 0 中部 オンライン 90.0 1 関東 オンライン 200.0 2 関東 店舗 100.0 3 関西 オンライン 80.0 4 関西 店舗 135.0
as_index=Falseというオプションを付けると、グループのキー(ここでは地域とチャネル)が列として残るので、結果が見やすくなります。
付けない場合は、これらのキーがインデックス(行の名前)になります。
なぜgroupbyが遅くなるのか
ここまでの例では、データが6行しかないので一瞬で処理が終わります。
しかし、実際の業務では数万行、数十万行のデータを扱うことが珍しくありません。そうなると、同じgroupbyでも処理に時間がかかるようになってきます。
なぜ遅くなるのでしょうか。
主な原因は4つあります。それぞれについて、原因と対策を見ていきましょう。
原因1:文字列のまま処理している
Pandasでは、「関東」「関西」のような文字列データは「object型」として扱われます。
コンピュータにとって、文字列の比較は数字の比較よりもずっと時間がかかる作業なのです。
たとえば、「関東」と「関西」が同じかどうかを判定するには、一文字ずつ比較する必要があります。これを何万回も繰り返すと、処理時間が長くなってしまいます。
そこで登場するのが「カテゴリ型(category型)」です。
カテゴリ型は、文字列を内部的に数字に変換して管理します。「関東=1、関西=2、中部=3」のように番号を振って、比較時は数字同士を比べるので高速になるのです。
# 文字列型(object)からカテゴリ型に変換
df["region"] = df["region"].astype("category")
df["channel"] = df["channel"].astype("category")
原因2:存在しないグループも処理している
カテゴリ型には「すべての可能な値のリスト」が記録されています。
たとえば地域のカテゴリに「北海道」が含まれていても、実際のデータには北海道のデータが1件もない、ということがあります。
通常のgroupbyでは、このような「存在しないグループ」も結果に含めようとするため、無駄な処理が発生します。
observed=Trueを指定すると、実際にデータに存在するグループだけを処理するようになります。
# 実際に存在するグループだけを処理 df.groupby(["region", "channel"], observed=True)["sales"].sum()
原因3:結果を並べ替えている
groupbyは初期設定では、グループのキーをアルファベット順(あいうえお順)に並べ替えます。
しかし、多くの場合、この並べ替えは必要ありません。
sort=Falseを指定すると、並べ替えをスキップして処理を高速化できます。
# 並べ替えをスキップ df.groupby(["region", "channel"], sort=False)["sales"].sum()
原因4:同じgroupbyを何度も実行している
売上の合計、平均、件数など、複数の集計値が必要な場合、ついつい別々にgroupbyを実行してしまいがちです。
しかし、これではグループ化の処理を何度も繰り返すことになり、非効率です。
# 非効率な例:3回もgroupbyを実行している
sum_result = df.groupby("region")["sales"].sum()
mean_result = df.groupby("region")["sales"].mean()
count_result = df.groupby("region")["sales"].count()
# 効率的な例:1回のgroupbyで複数の集計を実行
result = df.groupby("region")["sales"].agg(['sum', 'mean', 'count'])
aggは「aggregate(集約)」の略で、複数の集計を一度に実行できる便利な機能です。
実際に高速化を体験してみよう
理論はこのくらいにして、実際にどのくらい速くなるのか試してみましょう。
100万行のデータを使って、最適化前と最適化後の処理時間を比較します。
ステップ1:検証用データの準備
まず、実験用のデータを作成します。NumPyを使って、ランダムな売上データを生成します。
以下、コードです。
import numpy as np
import pandas as pd
import time
# 乱数のシードを固定
np.random.seed(42)
# 100万行のデータを生成
N = 1_000_000 # アンダースコアで区切ると読みやすい
regions = ["関東", "関西", "中部", "東北", "九州"]
channels = ["オンライン", "店舗", "パートナー", "電話"]
df = pd.DataFrame({
"region": np.random.choice(regions, size=N), # 地域をランダムに選択
"channel": np.random.choice(channels, size=N), # チャネルをランダムに選択
"sales": np.random.normal(500000, 10000, size=N).round(3) # 平均500000、標準偏差10000の正規分布
})
print(f"データのサイズ: {len(df):,}行") # カンマ区切りで表示
print(df.head()) # 最初の5行を確認
以下、実行結果です。
データのサイズ: 1,000,000行
region channel sales
0 東北 オンライン 497092.948
1 九州 パートナー 482762.557
2 中部 店舗 497356.177
3 九州 店舗 494995.646
4 九州 電話 490321.006
ステップ2:最適化前の処理速度を測定
まず、何も最適化していない状態で処理時間を測定してみます。
以下、コードです。
# データ型を確認(regionとchannelがobject型になっているはず)
print("現在のデータ型:")
print(df.dtypes)
print()
# メモリ使用量を確認
memory_mb = df.memory_usage(deep=True).sum() / 1024**2
print(f"メモリ使用量: {memory_mb:.2f} MB")
print()
# 速度を表示
start_time = time.perf_counter() # 正確な時間測定用のタイマーを開始
result_before = df.groupby(["region", "channel"])['sales'].agg(['sum', 'mean', 'count'])
time_before = time.perf_counter() - start_time
print(f"処理時間(最適化前): {time_before:.3f} 秒")
以下、実行結果です。
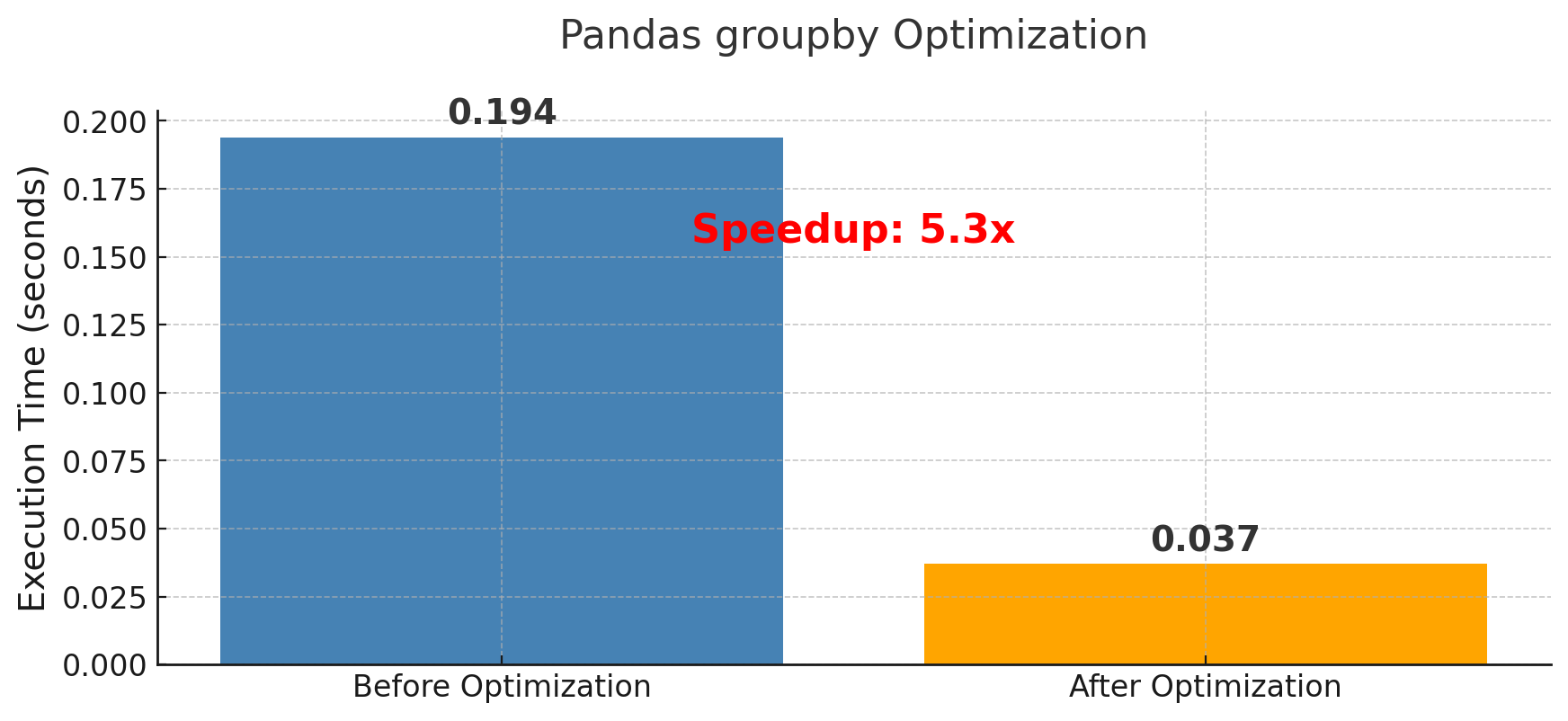
現在のデータ型: region object channel object sales float64 dtype: object メモリ使用量: 174.52 MB 処理時間(最適化前): 0.194 秒
私の環境では、0.194秒かかりました。
たった0.194秒と思うかもしれませんが、これが1日に何千回も実行される処理だったらどうでしょうか。
また、データがもっと大きくなったらどうでしょうか。小さな改善も積み重なれば大きな差になります。
ステップ3:データ型を最適化して再測定
それでは、先ほど学んだ3つのテクニックをすべて適用してみましょう。
以下、コードです。
# データをコピー(元のデータは残しておく)
df_optimized = df.copy()
# カテゴリ型に変換
df_optimized["region"] = df_optimized["region"].astype("category")
df_optimized["channel"] = df_optimized["channel"].astype("category")
print("最適化後のデータ型:")
print(df_optimized.dtypes)
print()
# メモリ使用量を再確認
memory_mb_optimized = df_optimized.memory_usage(deep=True).sum() / 1024**2
print(f"メモリ使用量: {memory_mb_optimized:.2f} MB")
print(f"メモリ削減率: {(1 - memory_mb_optimized/memory_mb)*100:.1f}%")
print()
# 最適化された処理の実行
start_time = time.perf_counter()
result_after = df_optimized.groupby(
["region", "channel"],
observed=True, # 実際に存在するグループだけを処理
sort=False # 並べ替えをスキップ
)['sales'].agg(['sum', 'mean', 'count'])
# 速度を表示
time_after = time.perf_counter() - start_time
print(f"処理時間(最適化後): {time_after:.3f} 秒")
print(f"高速化率: {time_before / time_after:.1f}倍")
以下、実行結果です。
最適化後のデータ型: region category channel category sales float64 dtype: object メモリ使用量: 9.54 MB メモリ削減率: 94.5% 処理時間(最適化後): 0.037 秒 高速化率: 5.3倍
多くの場合、3倍から10倍程度の高速化が実現できます。
また、メモリ使用量も50%以上削減されることが珍しくありません。
カテゴリ型が効果的かどうかを判定する方法
カテゴリ型への変換は魔法の杖ではありません。
すべての文字列列をカテゴリ型にすればよいわけではなく、効果的な場合とそうでない場合があります。
判定の基準は「ユニーク率」です。
ユニーク率とは、「異なる値の数÷全体の行数」で計算される値です。
ここでは、そのための関数を作成し実施します。
以下、コードです。
"""カテゴリ型への変換が効果的かどうかを判定する関数"""
def check_category_effectiveness(df, column_name):
# ユニーク値の数を数える
unique_count = df[column_name].nunique()
total_count = len(df)
unique_rate = unique_count / total_count
print(f"列名: {column_name}")
print(f"異なる値の数: {unique_count:,}個")
print(f"全体の行数: {total_count:,}行")
print(f"ユニーク率: {unique_rate:.2%}")
# 判定結果を表示
if unique_rate < 0.05:
print("→ カテゴリ型への変換を強く推奨します!")
print(" 理由: 同じ値が何度も繰り返されているため、")
print(" 番号での管理が非常に効率的です。")
elif unique_rate < 0.5:
print("→ カテゴリ型への変換をお勧めします。")
print(" 理由: ある程度の重複があるため、効果が期待できます。")
else:
print("→ カテゴリ型への変換は推奨しません。")
print(" 理由: ほぼすべての値が異なるため、")
print(" 番号管理のメリットが少ないです。")
return unique_rate
# 各列を分析
for col in df.columns:
check_category_effectiveness(df, col)
print() # 見やすくするための空行
以下、実行結果です。
列名: region
異なる値の数: 5個
全体の行数: 1,000,000行
ユニーク率: 0.00%
→ カテゴリ型への変換を強く推奨します!
理由: 同じ値が何度も繰り返されているため、
番号での管理が非常に効率的です。
列名: channel
異なる値の数: 4個
全体の行数: 1,000,000行
ユニーク率: 0.00%
→ カテゴリ型への変換を強く推奨します!
理由: 同じ値が何度も繰り返されているため、
番号での管理が非常に効率的です。
列名: sales
異なる値の数: 986,077個
全体の行数: 1,000,000行
ユニーク率: 98.61%
→ カテゴリ型への変換は推奨しません。
理由: ほぼすべての値が異なるため、
番号管理のメリットが少ないです。
地域名やチャネルのような、限られた値しか取らない列はカテゴリ型に最適です。
一方、顧客IDや注文番号のような、ほぼすべての行で異なる値を持つ列や、売上金額などの量的変数は、カテゴリ型には向きません。
大規模データを扱うときのテクニック
メモリに載りきらないような巨大なCSVファイルを処理する場合はどうすればよいでしょうか。
そんなときは「チャンク処理」という方法を使います。
チャンク処理とは、大きなファイルを小さな塊(チャンク)に分けて、少しずつ処理する方法です。
象を丸呑みすることはできませんが、一口サイズに切り分ければ食べられる、というイメージです。
今回は、先ほど作成したサンプルデータを使います。そのため、一旦CSVファイルとして保存します。
以下、コードです。
# デモ用にCSVファイルを作成
df.to_csv("sales_data.csv", index=False)
では、この巨大なCSVファイルを、チャンク処理で読み込みます。
そのための関数を作成し、それを使いこの巨大ファイルを読み込みます。
以下、コードです。
"""大規模CSVファイルを少しずつ読み込んで集計する関数"""
def process_large_csv(filename, chunksize=10_000):
print(f"ファイル '{filename}' を {chunksize:,}行ずつ処理します...")
# 集計結果を保存する変数(最初は空)
aggregated = None
chunk_count = 0
# ファイルを少しずつ読み込む
for chunk in pd.read_csv(filename, chunksize=chunksize):
chunk_count += 1
print(f" チャンク {chunk_count} を処理中...")
# 各チャンクでカテゴリ型に変換
chunk["region"] = chunk["region"].astype("category")
chunk["channel"] = chunk["channel"].astype("category")
# チャンクごとに集計
chunk_result = chunk.groupby(
["region", "channel"],
observed=True
)["sales"].sum()
# 結果を累積していく
if aggregated is None:
# 最初のチャンクの結果をそのまま保存
aggregated = chunk_result
else:
# 2番目以降は既存の結果に加算
# fill_value=0 により、片方にしかないキーも0として扱われる
aggregated = aggregated.add(chunk_result, fill_value=0)
print(f"処理完了!合計 {chunk_count} チャンクを処理しました。")
return aggregated
# チャンク処理を実行
result = process_large_csv("sales_data.csv", chunksize=20_000)
print("\n集計結果(上位5件):")
print(result.head())
以下、実行結果です。
ファイル 'sales_data.csv' を 20,000行ずつ処理します...
チャンク 1 を処理中...
チャンク 2 を処理中...
チャンク 3 を処理中...
チャンク 4 を処理中...
チャンク 5 を処理中...
チャンク 6 を処理中...
チャンク 7 を処理中...
チャンク 8 を処理中...
チャンク 9 を処理中...
チャンク 10 を処理中...
チャンク 11 を処理中...
チャンク 12 を処理中...
チャンク 13 を処理中...
チャンク 14 を処理中...
チャンク 15 を処理中...
チャンク 16 を処理中...
チャンク 17 を処理中...
チャンク 18 を処理中...
チャンク 19 を処理中...
チャンク 20 を処理中...
チャンク 21 を処理中...
チャンク 22 を処理中...
チャンク 23 を処理中...
チャンク 24 を処理中...
チャンク 25 を処理中...
チャンク 26 を処理中...
チャンク 27 を処理中...
チャンク 28 を処理中...
チャンク 29 を処理中...
チャンク 30 を処理中...
チャンク 31 を処理中...
チャンク 32 を処理中...
チャンク 33 を処理中...
チャンク 34 を処理中...
チャンク 35 を処理中...
チャンク 36 を処理中...
チャンク 37 を処理中...
チャンク 38 を処理中...
チャンク 39 を処理中...
チャンク 40 を処理中...
チャンク 41 を処理中...
チャンク 42 を処理中...
チャンク 43 を処理中...
チャンク 44 を処理中...
チャンク 45 を処理中...
チャンク 46 を処理中...
チャンク 47 を処理中...
チャンク 48 を処理中...
チャンク 49 を処理中...
チャンク 50 を処理中...
処理完了!合計 50 チャンクを処理しました。
集計結果(上位5件):
region channel
中部 オンライン 2.483739e+10
パートナー 2.504528e+10
店舗 2.502897e+10
電話 2.506240e+10
九州 オンライン 2.491706e+10
Name: sales, dtype: float64
このように、一度にすべてをメモリに載せることなく、大規模なデータを処理できます。
ちなみに、fill_value=0は重要なオプションです。あるチャンクには「関東×オンライン」のデータがあっても、別のチャンクにはない場合に、0として扱います。
さらに高速化するためのテクニック
ここまでの基本的な最適化でも十分な効果が得られますが、さらに高速化したい場合のテクニックを3つ紹介します。
それぞれ異なるアプローチで処理を効率化できるので、状況に応じて使い分けることが大切です。
テクニック1:ベクトル化による高速化
まず最初に紹介するのは「ベクトル化」という考え方です。
これは、グループごとに処理を繰り返すのではなく、全体に対して一度に処理を行ってからグループ化するというアプローチです。
具体例として、「売上が1200円を超える割合を地域ごとに計算したい」という場合を考えてみましょう。
まずは、ベクトル化を利用せずにapplyで実施します。
以下、コードです。
# データの準備
N = 200_000
df_large = pd.DataFrame({
"region": np.random.choice(regions, size=N),
"sales": np.random.normal(1000, 200, size=N).round(2)
})
# apply使用
def calculate_high_sales_ratio_slow(df):
"""各地域で売上1200超の割合を計算(遅い方法)"""
return df.groupby("region")["sales"].apply(
lambda x: (x > 1200).mean() # 各グループに対してPython関数を実行
)
# 速度を表示
start = time.perf_counter()
result_slow = calculate_high_sales_ratio_slow(df_large)
time_slow = time.perf_counter() - start
print(f"apply使用: {time_slow:.3f}秒")
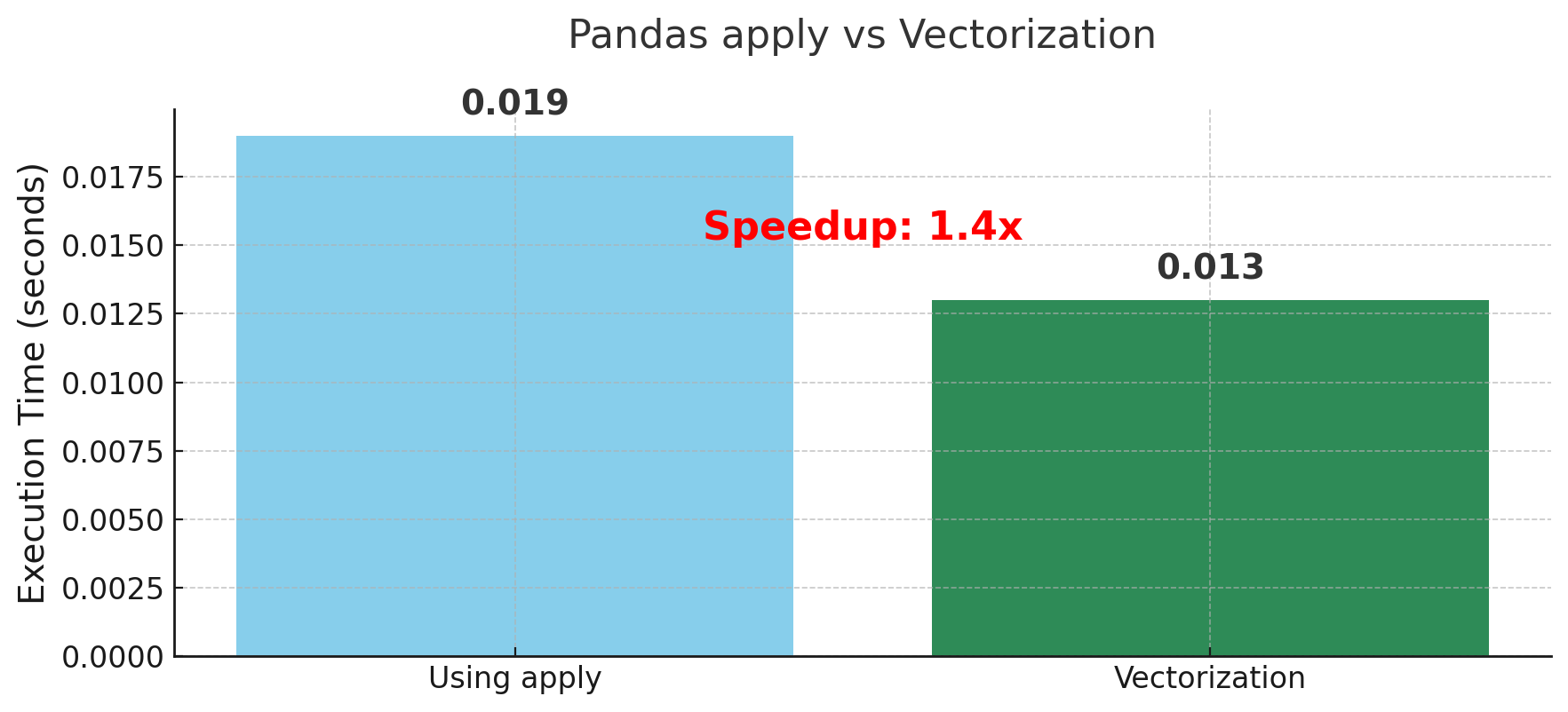
以下、実行結果です。
apply使用: 0.019秒
次に、ベクトル化して実施します。
以下、コードです。
# ベクトル化
def calculate_high_sales_ratio_fast(df):
# まず、売上が1200を超えるかどうかの列を作る
df_with_flag = df.copy()
df_with_flag['is_high_sales'] = df_with_flag["sales"] > 1200
# True/Falseの平均を取ると、Trueの割合が計算できる
return df_with_flag.groupby("region")["is_high_sales"].mean()
# 速度を表示
start = time.perf_counter()
result_fast = calculate_high_sales_ratio_fast(df_large)
time_fast = time.perf_counter() - start
print(f"ベクトル化: {time_fast:.3f}秒")
print(f"高速化率: {time_slow/time_fast:.1f}倍")
以下、実行結果です。
ベクトル化: 0.013秒 高速化率: 1.4倍
なぜベクトル化が速いのでしょうか。
applyを使った場合、Pandasは各グループ(ここでは5つの地域)に対して、それぞれPython関数を呼び出します。これは、Pythonレベルのループが5回実行されることを意味します。
一方、ベクトル化された処理では、まず全20万行に対して一度に「1200を超えるか」の判定を行い、その後でグループ化して平均を計算します。
NumPyの高速な配列処理を活用できるため、はるかに高速になるのです。
テクニック2:複数集計は1回で(Named Aggregation)
次に紹介するのは、複数の集計を効率的に行う方法です。
売上の合計、平均、最大値など、複数の値を計算したいとき、どのようにコードを書いていますか?
まず、よくある非効率な書き方を見てみましょう。
以下、コードです。
# 準備:数量(qty)列も追加したデータを作成
df_multi = pd.DataFrame({
"region": np.random.choice(regions, size=N),
"channel": np.random.choice(channels, size=N),
"sales": np.random.normal(1000, 200, size=N).round(2),
"qty": np.random.randint(1, 5, size=N) # 数量
})
# 非効率な方法:同じgroupbyを4回も実行
start = time.perf_counter()
sum_df = df_multi.groupby(["region", "channel"])['sales'].sum().rename('sales_sum')
mean_df = df_multi.groupby(["region", "channel"])['sales'].mean().rename('sales_mean')
qty_df = df_multi.groupby(["region", "channel"])['qty'].sum().rename('qty_sum')
size_df = df_multi.groupby(["region", "channel"]).size().rename('n')
# 結果を結合
result_bad = pd.concat([sum_df, mean_df, qty_df, size_df], axis=1).reset_index()
# 速度を表示
time_bad = time.perf_counter() - start
print(f"非効率な方法: {time_bad:.3f}秒")
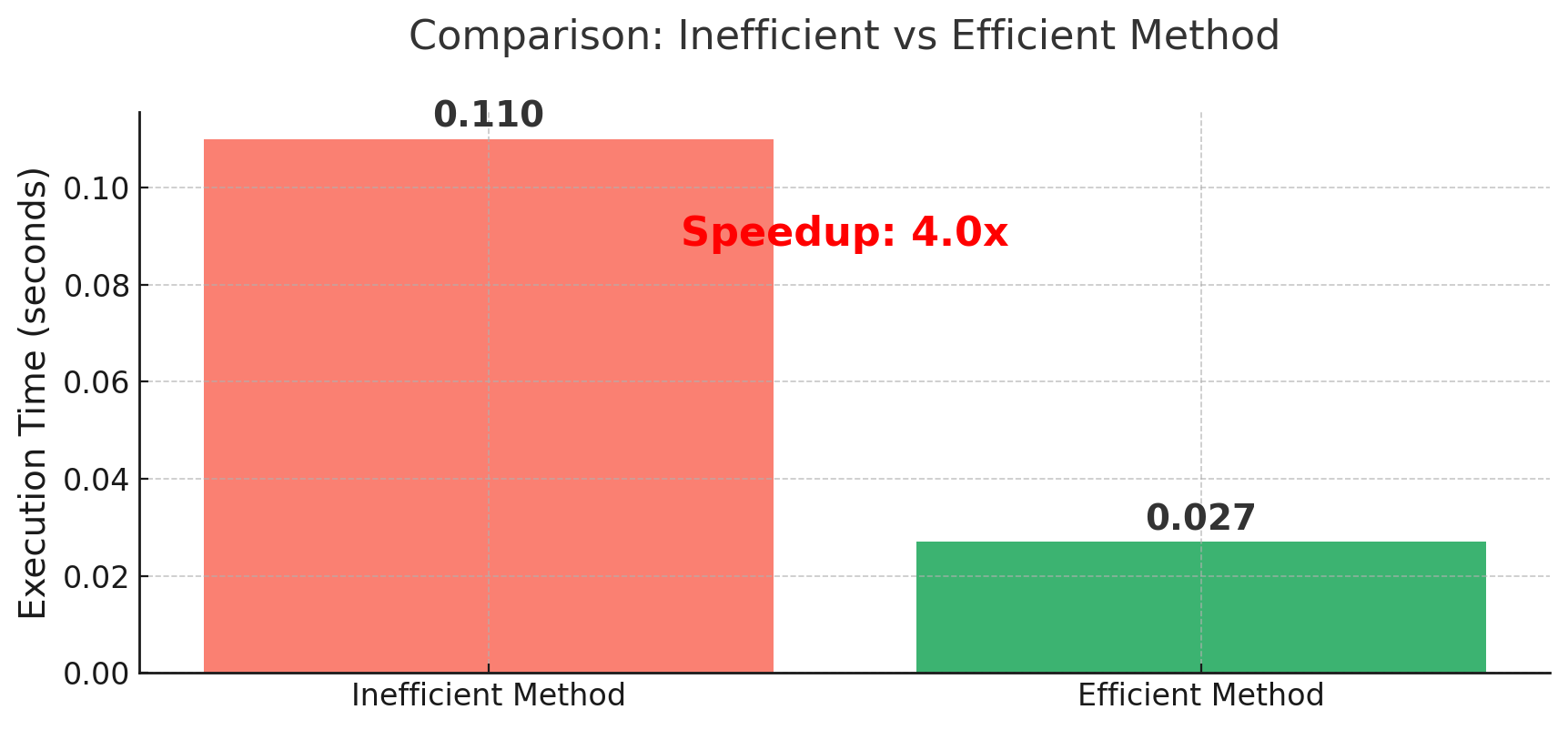
以下、実行結果です。
非効率な方法: 0.110秒
この書き方の問題点は、同じグループ化処理を4回も繰り返していることです。グループ化は意外と重い処理なので、これを何度も繰り返すのは無駄です。
そこで、Named Aggregationという機能を使って、1回のgroupbyですべての集計を行います。
以下、コードです。
# 効率的な方法:Named Aggregationで1回のgroupbyで完結
start = time.perf_counter()
result_good = (
df_multi.groupby(["region", "channel"], observed=True, sort=False)
.agg(
sales_sum=("sales", "sum"), # 売上の合計を sales_sum という名前で
sales_mean=("sales", "mean"), # 売上の平均を sales_mean という名前で
qty_sum=("qty", "sum"), # 数量の合計を qty_sum という名前で
n=("sales", "size"), # 件数を n という名前で
)
.reset_index()
)
# 速度を表示
time_good = time.perf_counter() - start
print(f"効率的な方法: {time_good:.3f}秒")
print(f"高速化率: {time_bad/time_good:.1f}倍")
以下、実行結果です。
効率的な方法: 0.027秒 高速化率: 4.0倍
Named Aggregationの書き方は、列名=(対象列, 集計関数)という形式です。
この方法を使うと、どの列に対してどんな集計を行うのかが一目でわかり、コードも読みやすくなります。
また、結果の列名も自由に指定できるので、後続の処理も書きやすくなります。
さらに、同じ列に対して複数の集計を行いたい場合も、簡潔に書けます。
以下、コードです。
# 売上列に対して複数の統計量を一度に計算
result_stats = (
df_multi.groupby("region", observed=True, sort=False)
.agg(
売上合計=("sales", "sum"),
売上平均=("sales", "mean"),
売上中央値=("sales", "median"),
売上最大=("sales", "max"),
売上最小=("sales", "min"),
件数=("sales", "count")
)
)
print(result_stats)
以下、実行結果です。
売上合計 売上平均 売上中央値 売上最大 売上最小 件数 region 関西 40215053.68 1000.573589 1001.020 1733.19 230.39 40192 中部 40078244.96 1000.455441 999.255 1908.88 152.53 40060 関東 39632871.78 999.568015 999.055 1829.39 210.63 39650 東北 40054378.43 999.410610 999.635 1905.63 224.68 40078 九州 39995762.98 999.394377 1000.035 1787.66 231.72 40020
テクニック3:PyArrowバックエンドの活用(Pandas 2.0以降)
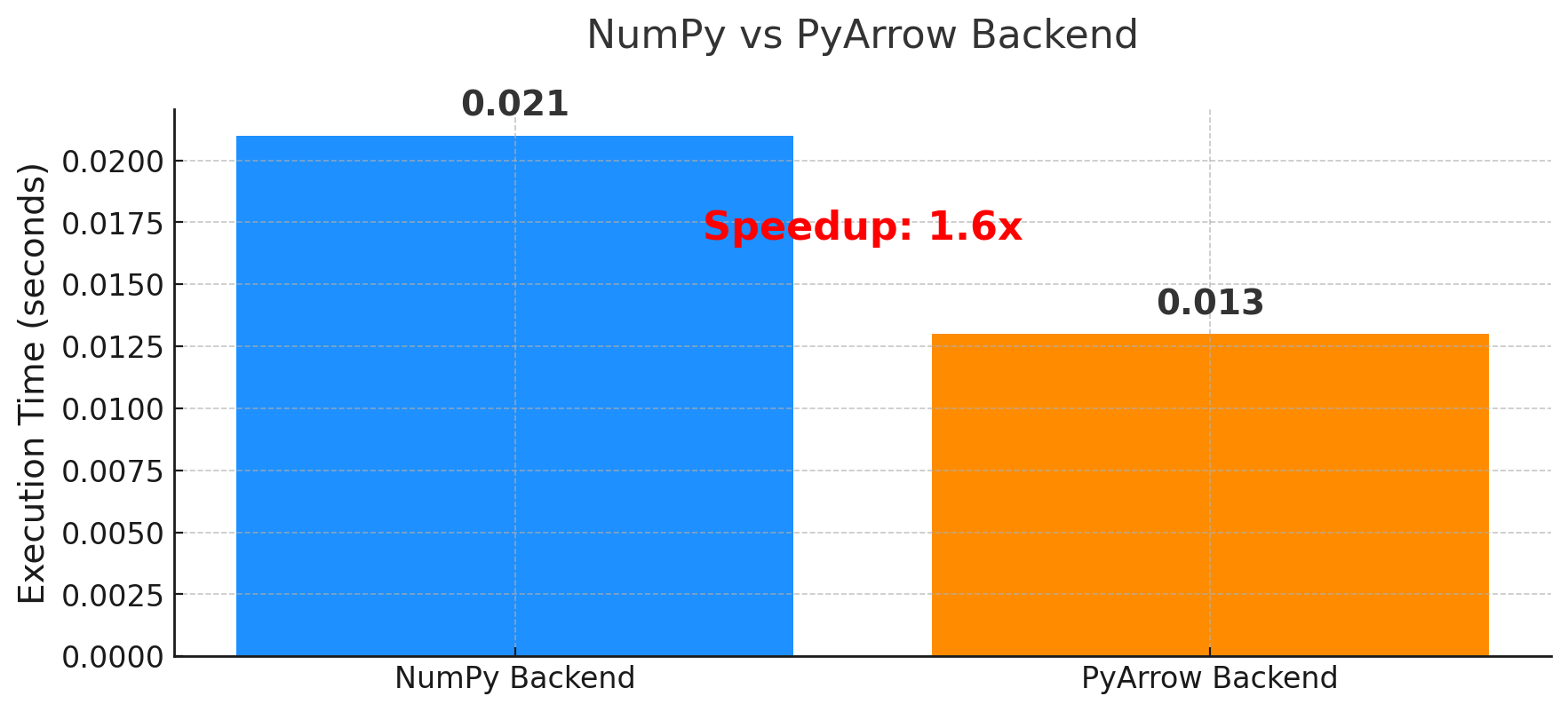
最後に紹介するのは、比較的新しい機能である「PyArrowバックエンド」です。
これは、Pandasの内部でデータを管理する方法を変更することで、処理を高速化する技術です。
従来、PandasはNumPyという数値計算ライブラリを使ってデータを管理していました。
しかし、Pandas 2.0からは、Apache Arrowという新しい形式でデータを管理することもできるようになりました。
特に文字列データや欠損値を含むデータの処理で、大きな性能改善が期待できます。
まず、従来のNumPyバックエンドで処理した場合です。
以下、コードです。
# 従来のNumPyバックエンドで処理
start = time.perf_counter()
result_numpy = df_multi.groupby(["region", "channel"], sort=False)["sales"].mean()
# 速度を表示
time_numpy = time.perf_counter() - start
print(f"NumPyバックエンド: {time_numpy:.3f}秒")
以下、実行結果です。
NumPyバックエンド: 0.021秒
次に、PyArrowバックエンドに変換し処理した場合です。
以下、コードです。
# PyArrowバックエンドに変換し処理
# dtype_backend="pyarrow"を指定することで、PyArrow形式に変換
df_arrow = df_multi.convert_dtypes(dtype_backend="pyarrow")
# 速度を表示
start = time.perf_counter()
result_arrow = df_arrow.groupby(["region", "channel"], sort=False)["sales"].mean()
time_arrow = time.perf_counter() - start
print(f"\nPyArrowバックエンド: {time_arrow:.3f}秒")
print(f"高速化率: {time_numpy/time_arrow:.1f}倍")
以下、実行結果です。
PyArrowバックエンド: 0.013秒 高速化率: 1.6倍
PyArrowバックエンドの利点は、高速化だけではありません。
欠損値の扱いがより直感的になり、メモリ効率も改善されます。
ただし、すべての機能が完全に互換性があるわけではないので、既存のコードを移行する際は動作確認が必要です。
まとめ
ここまで多くのテクニックを紹介してきましたが、最初から全部を覚える必要はありません。
まずは次の3つのステップから始めてみてください。
最初のステップは、現在使っているデータフレームのデータ型を確認することです。
df.dtypesを実行して、object型の列がないか確認してみましょう。
特に、地域名、カテゴリ名、ステータスなど、限られた値しか取らない列がobject型になっていたら、それは改善のチャンスです。
次のステップは、そのような列をカテゴリ型に変換することです。
df["列名"] = df["列名"].astype("category")という一行を追加するだけで、処理速度とメモリ効率が大幅に改善される可能性があります。
最後のステップは、groupbyを使うときにobserved=Trueとsort=Falseを指定することです。
さらに、複数の集計が必要な場合は、Named Aggregationを使って1回のgroupbyで済ませることを心がけましょう。
これらのオプションを付けるだけで、無駄な処理を省くことができます。
データ分析の世界では、このような小さな改善の積み重ねが、大きな差を生み出します。
最初は効果が小さく感じるかもしれませんが、データが大きくなったり、処理を繰り返したりする場面で、その違いを実感することでしょう。
今回紹介したテクニックは、どれも今すぐ試せるものばかりです。
まずは手元のコードでdf.dtypesを実行してみてください。そして、object型の列があったら、カテゴリ型への変換を試してみてください。
きっと、想像以上の改善が得られるはずです。

